はじめに
今回は次世代シークエンサー(next-generation sequencer; NGS)を用いたRNA-seq解析の基礎知識について解説します。はじめてRNAseq解析をはじめる方、途中で挫折してしまった方、公共のデータベースを用いて再解析したい方、などに読んでいただきたい初心者用の記事となっております。
私自身ウェット系ラボの出身であり、公共データベースを使って初めてドライ解析を始めた時はプログラミング以外の箇所でも非常に苦労しました。例えば、 次世代シークエンサーから出力される生データのデータ構造の複雑さ、 名前がよく似たアクセッション番号の違い、 解析に用いるファイル形式の種類、などです。これらの内容は解説書やネットで調べても、初心者向けの情報が乏しく、分かりづらいものも多かったので取り上げました。
どのような情報・知識があればドライ解析入門者がRNAseq解析に取り組みやすくなるかを重点的に考え、今回はNGSのデータ構造について丁寧に解説しています!ぜひご覧ください!
※次世代シークエンサーの種類とその原理、違いについては下記の記事で解説しています。
SRAとは
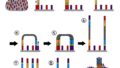
次世代シーケンシングの解析結果を論文に投稿する際は、そのデータをSRA(Sequence Read Archive)と呼ばれる公共データベースに登録することが義務付けられています。SRAとは次世代シークエンサーが出力した生データのリポジトリのことで、 米国のNCBI(National Center for Biotechnology Information)が運営しています。SRAには日本版と欧州版があり、日本版をDDBJ Sequence Read Archive(DRA)、欧州版をEBI Sequence Read Archive(ERA)と言います。SRA、DRA、ERAは名前こそ若干異なりますが、中身のデータ構造は同じです。DRAは日本のDDBJ(DNA Data Bank of Japan)が運営し、ERAは欧州のEBI(European Bioinformatics Institute)が運営しています。研究者は論文投稿時にNCBI/DDBJ/EBIのいずれかのデータベースに生データを登録し、そのデータに与えられたアクセッション番号を引用して論文を投稿します。 NCBI/DDBJ/EBIの各拠点に集まったデータはミラーリングされており、 論文に記載のアクセッション番号を使って、いずれの拠点のデータベースからでも目的のデータにアクセスすることができます。
NCBI/DDBJ/EBIの3つに分かれているのはアクセス負荷分散やデータのバックアップ等の意味があり、 NCBI/DDBJ/EBIの国際的な協力機構をInternational Nucleotide Sequence Database Collaboration(INSDC)と呼びます(図1)。
本体のデータを説明する情報(作成者、日付、保存場所など)が記載されたデータのこと。
貯蔵庫、倉庫、保存容器という意味の単語。メタデータが格納されている場所を示す。
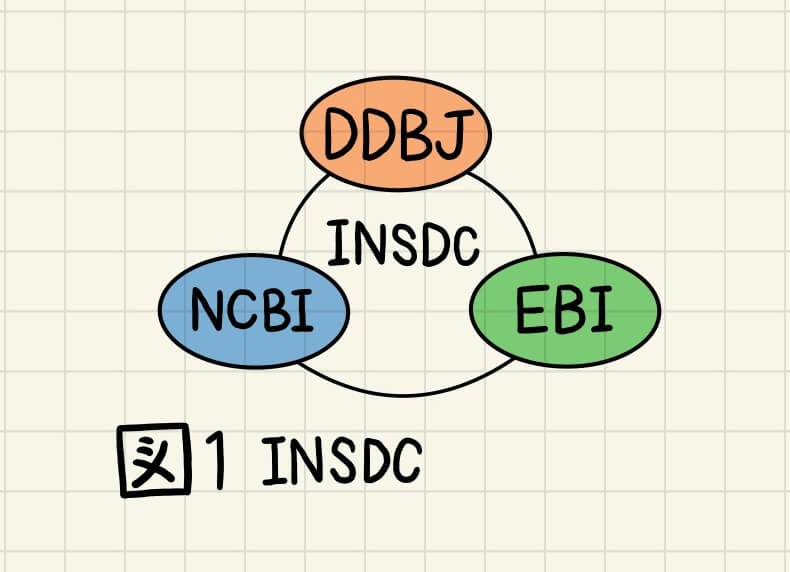
SRAファイルのデータ構造
それではまずSRAファイルのデータ構造について解説します。なぜ最初にデータ構造を解説するかというと、様々な種類のデータが似たような名前のアクセッション番号によって管理されており、自分が調べたいデータがどのアクセッション番号のデータに格納されているかわかりづらいからです。
SRAメタデータは相互に関連している複数のオブジェクト(Submission、BioProject、Analysis、Experiment、BioSample、Run)から構成されています(図2)。
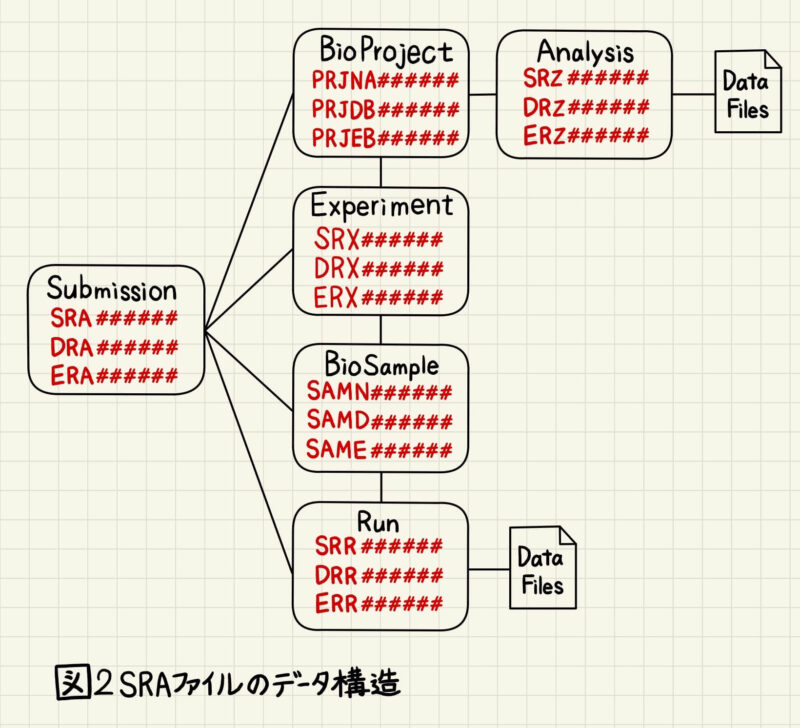
Submission、BioProject、Analysis、Experiment、BioSample、RunにはそれぞれSRA、PRJNA、SRZ、SRX、SAMN、SRRから始まるアクセッション番号が付与されています。DDBJに登録されたDRAメタデータの場合は、各オブジェクトにDRA、PRJDB、DRZ、DRX、SAMD、DRRというアクセッション番号が付与されます。同様に、EBIに登録されたERAの場合は各オブジェクトにERA、PRJEB、ERZ、ERX、SAME、ERRが付与されます。Submission、BioProject、Analysis、Experiment、BioSample、Runに格納されているデータは下記の通りで、NGSから出力されたデータはこのように整理されて各オブジェクトに格納されています。
- Submission(SRA/DRA/ERA#######)はBioProject、Analysis、Experiment、BioSample、Runデータを格納した一番上の階層のフォルダです。
- BioProject(PRJNA/PRJDB/PRJEB#######)は研究のタイトル、アブストラクト、論文のリンク、登録者名、登録者の所属機関名、解析に用いたサンプル名、解析結果などが格納されています。
- Analysis(SRZ/DRZ/ERZ#######)はBioProjectを解析したデータが格納されています。Analysisの登録は任意であり、INSDCで共有されません。
- Experiment(SRX/DRX/ERX#######)は解析に用いた次世代シークエンサーの機器名、ライブラリの調製方法、リード数などのRUN情報が格納されています。1つのSRX/DRX/ERXに対して、1つのSAMN/SAMD/SAMEデータおよび1つのSRR/DRR/ERRデータが存在します。
- BioSample(SAMN/SAMD/SAME######)にはサンプル情報が格納されています。
- Run(SRR/DRR/ERR######)はリード数やリード長などのシーケンスデータが格納されています。
BioProjectとBioSampleはそれぞれ2011年、2014年に運用を開始したシステムで、 従来のSRAファイルはBioProjectの代わりにStudy(SRP/DRP/ERP######)、BioSampleの代わりにSample(SRS/DRS/ERS######)というデータベースが使用されていました(図3)。したがって、上述の6つのデータに加えて、StudyとSampleのアクセッション番号が付与されているSRAファイルもあります。BioProjectとStudy、BioSampleとSampleは大体同じ内容が記載されています。
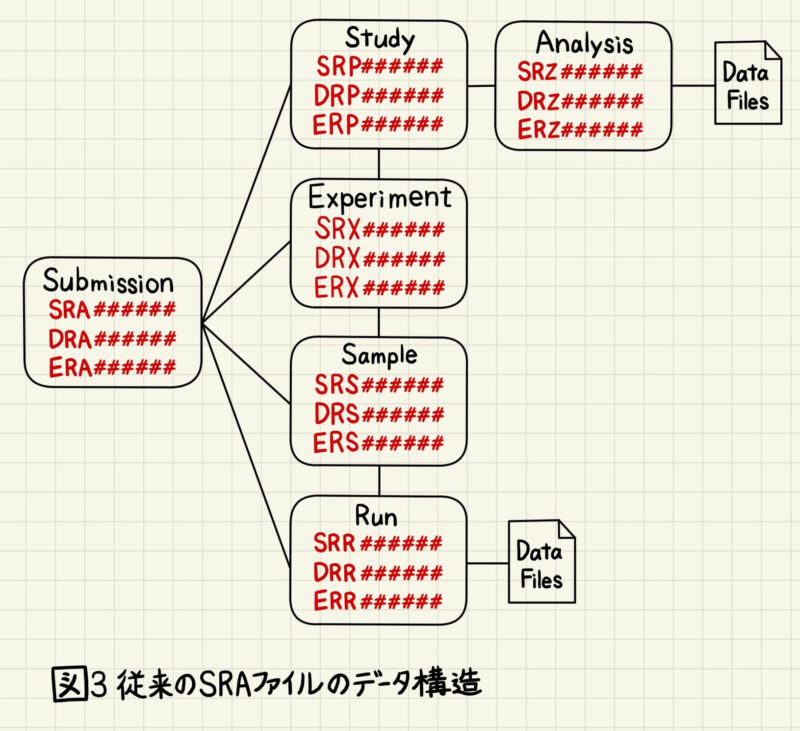
なぜStudyとSampleというデータベースがあるのに、新しくBioProjectとBioSampleが新設されたかというと下記の理由があります。
従来、遺伝子発現解析はマイクロアレイを用いて行われ、マイクロアレイから得られた遺伝子発現解析データはNCBIのGEO(Gene Expression Omnibus)に保存されていました。しかし、NGS技術の登場により爆発的にデータ量が増え、増え続けるNGSのデータに対応するため2008年にSRAが作られました。現行ではGEOにもNGSの遺伝子発現解析データが入ることになっており、このままでは同一実験由来のデータが異なるデータベースに散在してしまい、データの再利用性が低下してしまいます。そこで、データの再利用性を向上させるため、異なるデータベース間で共通のアクセッション番号が作成されました。これがBioProjectとBioSampleです。

アクセッション番号の一文字目がSであればNCBIに、DであればDDBJに、EであればEBIに登録されたデータであることがわかります。また、アクセッション番号の三文字目を見れば観覧しているデータにどのような情報が記載されているかわかります。
GEO、GEA、ArrayExpressとは
前項で登場したGEOは、マイクロアレイやNGSなどの機能ゲノミクスデータを保存しているデータベースです。GEOはPlatfrom、Samples、Seriesから構成されています。Platfrom、Samples、SeriesにはそれぞれGPL、GSM、GSEから始まるアクセッション番号が付与されており、格納されている情報は下記の通りです。
- Platfrom(GPL#####)には実験に用いたマイクロアレイ、次世代シークエンサーの機器情報などが格納されています。
- Samples(GSM#####)には実験目的、サンプル調製方法、実験条件、発現量データなどが格納されています。
- Series(GSE#####)は関連するサンプルをグループ化したものです。一部のSeriesはGEOによりキュレーションが行われ、DataSet(GDS#####)が作成されています。
GEOの他に遺伝子発現データを保存しているデータベースがあります。DDBJが運営しているGEAとEBIが運営しているArrayExpressです。
GEAもマイクロレイやNGSのデータを保存しており、 Experiment情報はE-GEAD-n、Array design情報はA-GEAD-nというアクセッション番号が付与されて保存されています。
ArrayExpressはマイクロアレイのデータを格納するデータベースであり、 Experiment情報はE-XXXX-n、Array design情報はA-XXXX-nというアクセッション番号が付与されて保存されています。(nは数字、XXXXは4文字のコードを表す)

データベースの種類が多くて、混乱してしまいますよね。。。
他にも塩基配列データであれば、NCBI GenBankやDDBJ、ENAなどがあります。
インターネット上の情報を収集・編集することで新しい価値を付与し、それを共有すること。
まとめ
- 次世代シークエンサー(next-generation sequencer; NGS)の生データはSRA/DRA/ERAに保存される
- SRA、DRA、ERAはそれぞれ米国のNCBI、日本のDDBJ、欧州のEBIが運営している
- NGSの登場以前は、マイクロアレイから得られる遺伝子発現情報はGEOに保存されていた
- GEO、GEA、ArrayExpressに保存されている遺伝子発現情報もある
- Kodama Y, Shumway M, Leinonen R; International Nucleotide Sequence Database Collaboration. The Sequence Read Archive: explosive growth of sequencing data. Nucleic Acids Res. 2012 Jan;40(Database issue):D54-6. doi: 10.1093/nar/gkr854. Epub 2011 Oct 18. PMID: 22009675; PMCID: PMC3245110.
- Alnasir J, Shanahan HP. Investigation into the annotation of protocol sequencing steps in the sequence read archive. Gigascience. 2015 May 9;4:23. doi: 10.1186/s13742-015-0064-7. PMID: 25960871; PMCID: PMC4425880.
- DDBJ. “Sequence Read Archive”. https://www.ddbj.nig.ac.jp/dra/submission.html
- EBI. “BioStudies”. https://www.ebi.ac.uk/biostudies/arrayexpress/help#ae-data
- Kodama Y, Mashima J, Kosuge T, Ogasawara O. DDBJ update: the Genomic Expression Archive (GEA) for functional genomics data. Nucleic Acids Res. 2019 Jan 8;47(D1):D69-D73. doi: 10.1093/nar/gky1002. PMID: 30357349; PMCID: PMC6323915.