はじめに
本記事では代表的な次世代シークエンサーの種類とその原理についてわかりやすく、そして詳しく解説して行きます。次世代シークエンスを扱う上で必須の知識や用語についても同時に解説していきます。次世代シークエンサーを用いて実験をしたい方やNGSデータ解析をしたい方などに役立つ内容となっております。ぜひ最後までご覧ください!
※公共データベースに蓄積されたシークエンス情報を利用して、RNA-seq解析を行うために必要な知識を下記の記事で詳しく解説しています。
※RNA-seq解析(ドライ解析)のワークフローは下記の記事で詳しく解説しています。合わせてご覧ください。
次世代シークエンサーのプラットフォーム
次世代シークエンス(Next-Generation Sequencing; NGS)は、1回のランで超並列的に数百万から数兆の塩基配列を決定することのできる技術です。名前に”次世代”とついていますが、2005年に次世代シークエンサーが登場してから既に15年以上経過しており、”次世代”という名称はもはや適していないという意見が出てきています。しかし、次世代シークエンスという呼び方は変わっておらず、本記事でもハイスループットに塩基配列を決定する技術を次世代シークエンスと表記します。
表1に代表的な次世代シークエンスプラットフォームをまとめました。これらのプラットフォームにはそれぞれ長所、短所があるのですが一つずつ解説していきます。その中でもIllumina社の次世代シークエンサーは2018年時点で市場シェアが約90%と圧倒的なシェアを誇っており、その原理を専門用語とともに特に詳しく解説していきます。

現在はIllumina社一強という状況ですが、次世代シークエンサーの開発競争は非常に激しく、Illumina社以外のメーカーもどんどん存在感を増してきています。
表1 代表的な次世代シークエンサーのプラットフォーム
| メーカー | Illumina | Pac Bio | Oxford Nanopore Technologies |
| 機種 | NovaSeq | Sequel II | MinION |
| Run Time | ~44hr | 30hr/SMRT cell | ~72hr |
| リード長 | 2 × 250 bp | ~15 kb | 4 Mb |
| 出力 | ~6000 Gb/run | ~500 Gb/run | ~50 Gb/run |
| エラー率 | – | 13-15% | 5-13% |
| 平均精度(Qスコア) [1] | 99.92% (Q31) | 99.95% (Q33) | 99.26% (Q21) |
| 長所 | ・ハイスループット ・出力あたりのコストが低い ・高精度 ・エラー率が低い | ・リード長が長い ・エピゲノム解析が可能 ・リアルタイムでデータ取得が可能 | ・機器のサイズが小さい ・機器価格が安い ・リアルタイムでデータ取得が可能 |
| 短所 | ・リード長が短い ・機器価格が高い | ・スループットが低い ・エラー率が高い ・低出力 | ・精度が低い ・エラー率が高い ・低出力 |
[1] Qスコア; 読み取った塩基の精度を示す。Q10で90%、Q20で99%、Q30で99.9%の精度を表す。次世代シークエンスにはQ30以上のスコアが求められる。塩基の読み取り精度は100%ではないので、同じ領域を繰り返しシークエンスして精度を高めるということが行われる。このシークエンスの回数を被覆率(coverage)といい、一般的に30回被覆される。
Illumina
まずはIlluminaシークエンサーの原理について詳しく解説していきます。
ライブラリ調製
ライブラリ調製のやり方は各メーカーのライブラリ調製キットによって若干異なるのですが、ここではベーシックなライブラリ調製法をご紹介します。
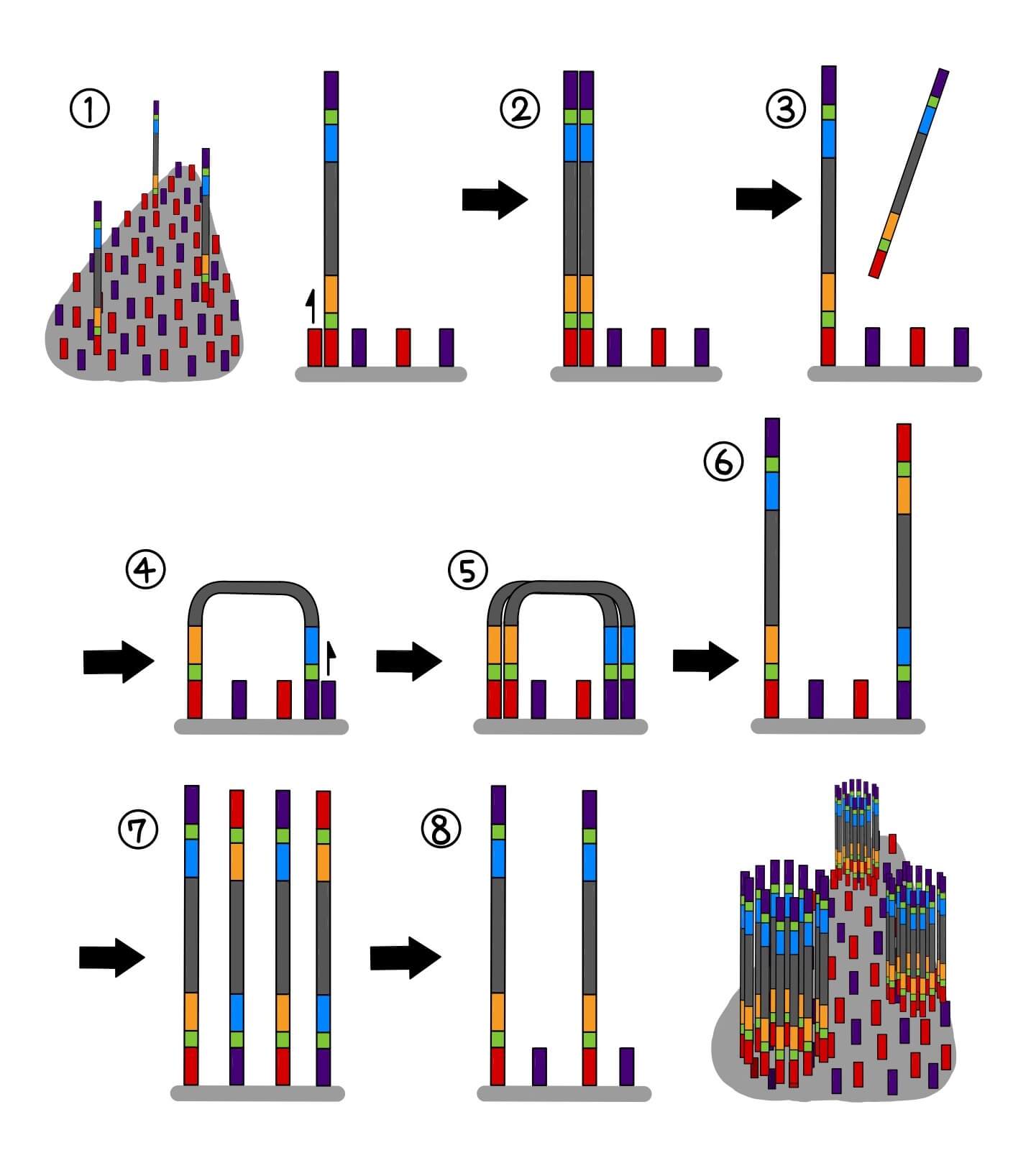
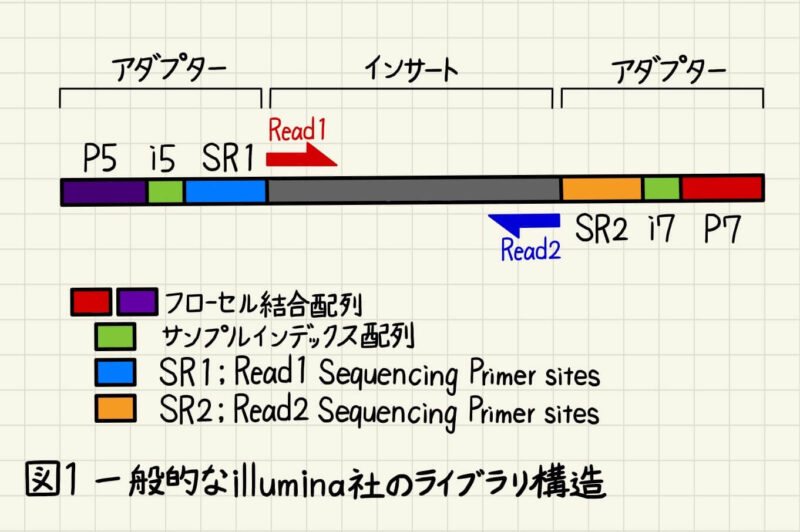
まずは対象のDNAまたはRNAを超音波処理や酵素処理により300-500bp程度の短い断片(フラグメント)にします。そしてフラグメントの両端にアダプターをライゲーションします(図1)。 アダプターにはフローセルというガラス基盤に結合するための配列(P5、P7と呼ばれます)、サンプルを識別するための配列(P5側のインデックス配列はi5、P7側のインデックス配列はi7と呼ばれます)、シークエンスプライマーが結合する配列(P5側の配列はSR1、P7側の配列はSR2と呼ばれます)が含まれています。アダプターに挟まれたDNAをインサートと言い、インサートにアダプターが付加されたフラグメントの集まりをライブラリと言います。また、ライブラリを作製するステップをライブラリ調製と呼びます。

サンプルごとに異なるインデックス配列を付与することで、シークエンスデータがどのサンプル由来かを判別できるようになり、複数サンプルを同じレーンで同時にシークエンスすることができます。
クラスタリング
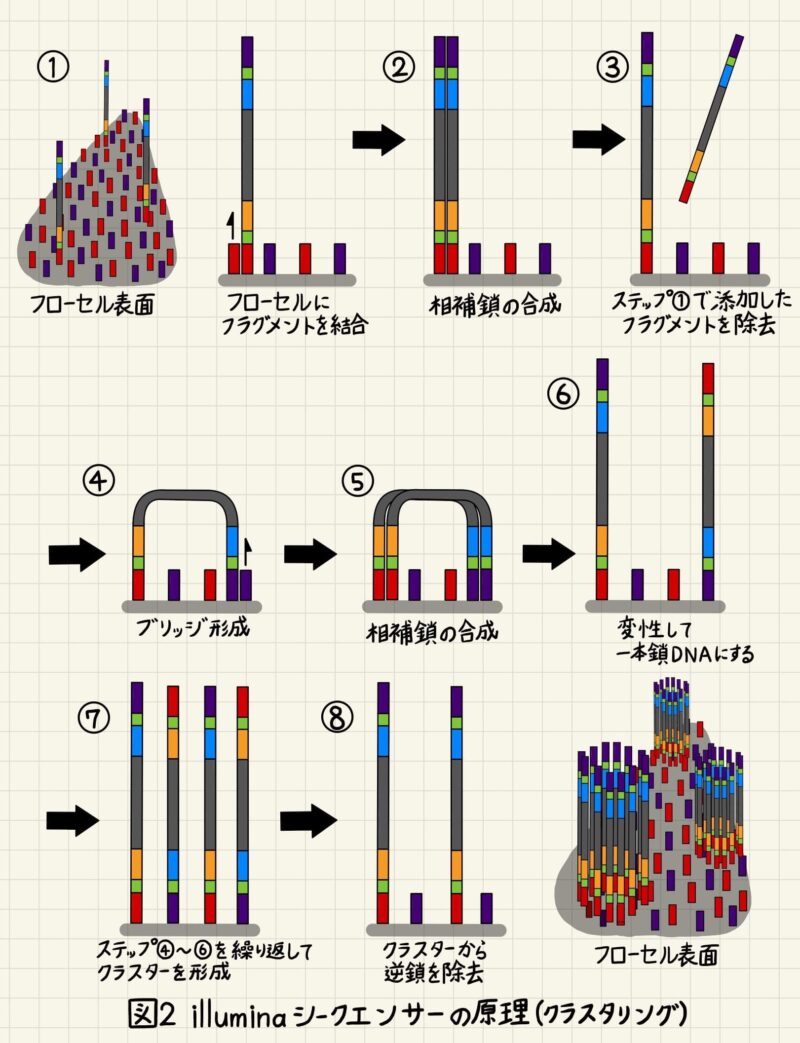
フローセルの表面には、フラグメントのフローセル結合配列に相補的な2種類の一本鎖DNAが高密度に配置されており、図2①のようにフラグメントと結合することができます。そしてフラグメントを鋳型としてDNAポリメラーゼを用いて相補鎖を合成します(図2②)。最初に添加したフラグメントは温度を上げて取り除きます(図2③)。新しく合成された一本鎖DNAは、フローセルに結合していない反対側のアダプターを使ってフローセルに結合します(図2④)。そして、橋のような構造を保ったまま、再び相補鎖を合成します(図2⑤)。その後、温度を上げて1本鎖DNAにします(図2⑥)。図2④~⑥のステップを繰り返すことで、2種類の方向性を持った一本鎖DNAのクラスターが形成されます(図2⑦)。最後に逆鎖を外し、順鎖のみを残します(図2⑧)。このクラスター作製方法は橋(ブリッジ)のような構造で伸長反応を繰り返すので、ブリッジPCRと呼ばれます。ブリッジPCR終了後は、フローセル上に順鎖のみから成るクラスターが形成されます(図2⑧右)。
シークエンス
IlluminaシークエンサーはDNAの伸長反応を進めながら塩基配列を決定するSequence-By-Synthesis(SBS)法という方法を採用しているので、まずはSBS法について見ていきましょう。
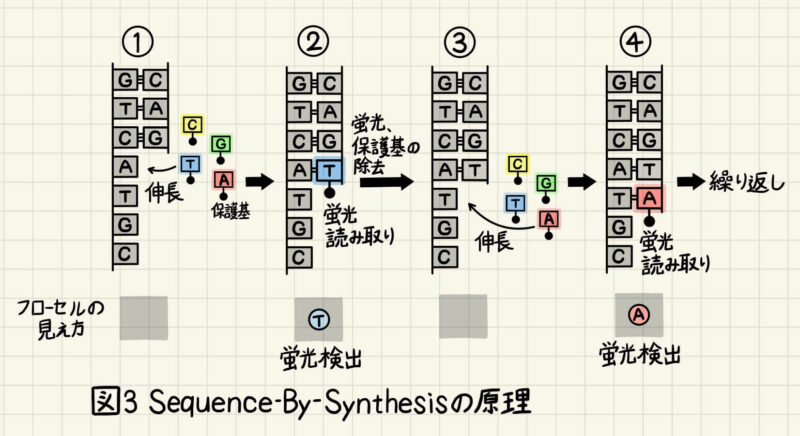
SBS法では可逆性の蛍光色素および保護基が結合した核酸を用いて相補鎖を合成します(図3①)。この核酸は3’末端に保護基がついているため、1塩基分だけ合成されたのち伸長反応は停止します。伸長が停止している間にレーザーを当てて蛍光を検出し、塩基を読み取ります(図3②)。その後、蛍光色素および保護基を取り除き、再び蛍光色素および保護基が結合した核酸を用いて相補鎖を合成します(図3③、④)。このステップを繰り返すことで、塩基配列を決定していきます。
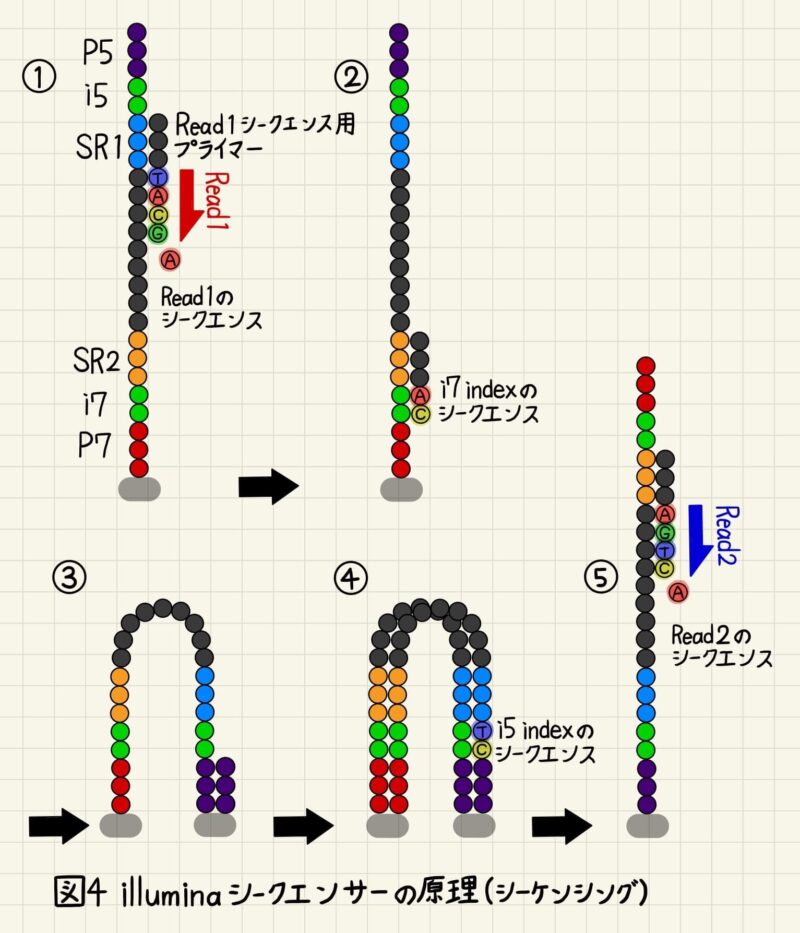
次はいよいよフローセルに固定化されたフラグメントのシークエンス原理について見ていきます。まずはRead1シークエンス用プライマーをSR1にアニーリングし、P5側のインサート末端の塩基配列(Read1)を読み取っていきます(図4①)。リード(Read)とはシークエンスで読み取られた配列データのことを言います。Illuminaシークエンサーはシークエンス反応が進むにつれ蛍光のバックグラウンドが上昇してしまうため、連続で読み取ることができる塩基の長さ(リード長)は150~250bp程度と短いのが特徴です。リード長が短いのでde novoアセンブリ等には適していません。一方で、高精度・高出力・出力あたりのコストの低さが長所のシークエンサーです。その後、相補鎖は取り除かれ、同様の方法でインデックス配列 i7の塩基配列を決定します(図4②)。
次に逆鎖を合成し、P7側のインサート末端の塩基配列(Read2)を読み取ります。この時インデックス配列 i5は逆鎖を合成するステップで読み取ります(図4④、⑤)。このようにインサートの両端をシークエンスする手法をペアエンド、インサートの片側のみをシークエンスする手法をシングルエンドと言います。ペアエンドはシングルエンドと比較するとデータ量が2倍になりますが、全体のリード長が増え、精度も向上するので、ほとんどの場合ペアエンドシークエンスが選択されます。表1中の”2 × 250 bp”という表記は、ペアエンドでインサートの末端から250bpをシークエンスするという意味になります。
Pac Bio
Pacific Biosciences(PacBio)社のシークエンスシステムはsingle-molecule-real-time (SMRT) sequencingと呼ばれており、Illumina社と比較してリード長が非常に長いことが特徴です(表1)。 Illuminaシークエンサーから出力される比較的短いリードはショートリードと呼ばれ、PacBio社や次項で紹介するOxford Nanopore Technologies社のように長いリードはロングリードと呼ばれます。リード長が非常に長いので、Illuminaシークエンサーが苦手としているde novoアセンブリや繰り返し配列の解読などの使用に適しています。 さらにPacBioシークエンサーは精度も高く、PacBio社の高精度(>99.9%の正確性)のロングリードはHiFi リードと呼ばれます。
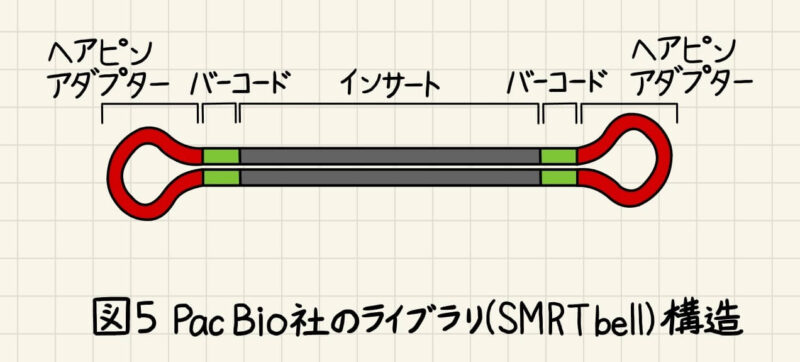
それではPacBio社のシークエンス原理について解説していきます。Bac Bio社のライブラリ調製は、Illumina社と異なり二本鎖DNAからスタートします。二本鎖DNAにサンプルを識別するバーコード配列とヘアピンアダプターを付加して図5のように環状構造の一本鎖DNAを作ります。このBac Bio社のライブラリをSMRTbellと言います。
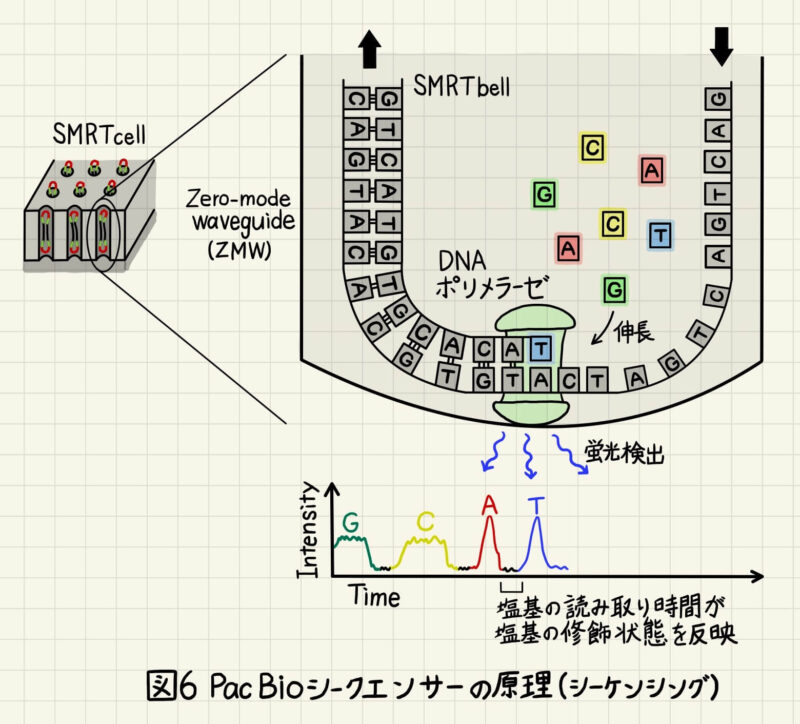
続いて、数千個のzero-mode waveguide (ZMW)が敷き詰められたSMATcellと呼ばれるシリコンチップにSMRTbellを添加します。各ZMWには単一のDNAポリメラーゼが固定されており、SMRTbellは円を描くように移動しながら複製されます(図6)。複製には蛍光色素標識核酸が使われており、核酸がDNAポリメラーゼに保持されたときの蛍光をZMWで検出します。また、塩基の取り込みの時間は塩基の修飾状態を反映しており、N6-methyladenine (m6A) や N4-methylcytosine (m4C)などの塩基修飾を直接検出することができます。このように環状DNAを繰り返しシークエンスする方法をCircular Consensus Sequencing(CCS)と言います。
しかしながら、 スループットの低さ、高いエラー率、出力あたりのコストの高さが短所です。
Oxford Nanopore Technologies
Oxford Nanopore Technologies社のシークエンサーはPacBio社よりもさらにリード長が長く、機器が手のひらに収まるほど小さいのが特徴です。また、シークエンスの原理はこれまでの方法と大きく異なり、DNAポリメラーゼを使用しません。
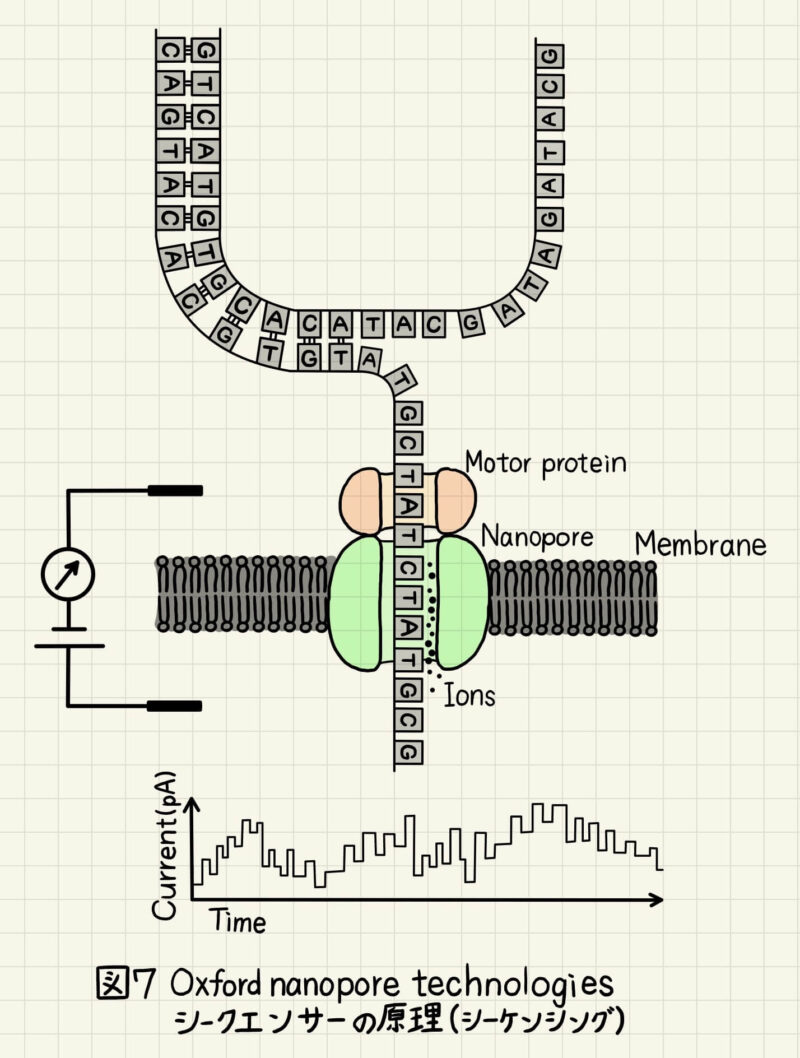
まず対象の二本鎖DNAにMotor proteinの結合部位となるアダプター配列を付与します。Motor proteinは二本鎖DNAを一本鎖DNAに解離し、一本鎖DNAを膜に埋め込まれたNanoporeに通す役割があります(図7)。膜には電圧がかけられてイオンの流れが発生しているのですが、一本鎖DNAの通過によりイオンの流れが妨げられ、Nanoporeでのイオン流入量が減少します。このイオンの流入量は各ヌクレオチドによって特有な値をとるため、塩基配列を読み取ることができます。また、メチル化修飾なども電流の変化として検出することができ、エピゲノム解析にも使用することができます。
リード長は圧倒的に長いですが、読み取り精度の低さや高いエラー率が短所です。

サンガーシーケンス法を第1世代として、Illuminaシークエンサーを第2世代、PacBioシークエンサー、Oxford Nanopore Technologieシークエンサーを第3世代と呼称する場合もあります。
まとめ
- 次世代シークエンサーのメーカーとしてIllumina、Pac Bio、Oxford Nanopore Technologiesが有名で、特にIlluminaシークエンサーは圧倒的なシェアを誇る。
- Illuminaシークエンサーはリード長が短いが、高精度でハイスループットでシークエンス可能。
- Pac Bioシークエンサーは低出力でエラー率が高いが、リード長が長い。
- Oxford Nanopore Technologiesシークエンサーは精度が低いが、リード長が非常に長い。
- Lee H, Min JW, Mun S, Han K. Human Retrotransposons and Effective Computational Detection Methods for Next-Generation Sequencing Data. Life (Basel). 2022 Oct 12;12(10):1583. doi: 10.3390/life12101583. PMID: 36295018; PMCID: PMC9605557.
- Loka, Tobias Pascal. Advanced Strategies for Alignment-based Real-time Analysis and Data Protection in Next-Generation Sequencing. Diss. 2020.
- PacBio. “WHOLE GENOME SEQUENCING”. https://www.pacb.com/products-and-services/applications/whole-genome-sequencing/