はじめに
mRNAの塩基配列情報を用いてタンパク質が合成されることを翻訳と言います。タンパク質はアミノ酸が鎖状につながったものなので、mRNAの塩基配列はアミノ酸配列を指定しているということになります。
DNA複製もRNAの転写もヌクレオチドを鋳型として新しいヌクレオチドを合成します。しかし、翻訳ではヌクレオチドであるmRNAから、構造的にヌクレオチドとは全く異なるタンパク質が合成されます。よく考えると不思議ですよね。 どうやってmRNAからタンパク質を合成しているのでしょうか?今回はこの翻訳機構、特にmRNAの遺伝情報がアミノ酸に変換される過程について詳しく解説していきます。
翻訳の仕組みはとてもよくできていて面白いです!今回も楽しく学んでいきましょう!
【難易度】★★★☆☆
【重要度】★★★★★
※RNAの基本知識は以下の記事で詳しく解説しています。
※mRNAの転写機構やRNA修飾については以下の記事で詳しく解説しています。
コドン
まずタンパク質の合成においてとても重要な概念である「コドン」について解説していきます。
mRNAは連続した3塩基で一つのアミノ酸を指定しています(表1)。このアミノ酸を指定する3塩基をコドンと言います。なぜ3塩基で一つのアミノ酸を指定しているかというと、RNAはA、C、G、Uの4種類で構成されているのに対し、タンパク質の構成要素であるアミノ酸は20種類もあるからです。実際にコドンの組み合わせを計算すると、「 4 × 4 × 4 = 64通り 」となり、3塩基あれば20種類あるアミノ酸を指定するのに十分であることがわかります。
しかし、アミノ酸は20種類しかないのに、コドンは64種類まで対応できるという点が気になりますよね。実は1つのコドンが1つのアミノ酸を指定しているのではなく、複数のコドンが一つのアミノ酸を指定しています。すなわち、64種類のコドンは全て、何らかのアミノ酸または終始コドンを指定しているということです。(終始コドンについては後述します)実際に表1のコドンとアミノ酸の対応表を見ると、1~6種類のコドンが1つのアミノ酸を指定していることが分かります。このコドンとアミノ酸の対応はほとんどの生物で共通しています。
また、「AUG」は開始コドンとしての役割もあり、mRNAの翻訳は必ずメチオニンで始まります。翻訳の終了を示すコドンは終始コドンと呼ばれ、表1のように「UAA」、「UAG」、「UGA」の3種類あります。終始コドンは3種類あるので、アミノ酸を指定するコドンは61種類となります。

タンパク質の翻訳では、「mRNAの3塩基分の遺伝情報 → 1つのアミノ酸」のように遺伝情報が移動するわけですね。

読み枠(リーディングフレーム)
mRNAの3塩基で1アミノ酸を指定しているので、mRNAの読み取られ方は一方向に3通りあります(図1)。二本鎖DNAのうち、どちらのDNA鎖もmRNAの鋳型鎖として使われる場合があるので、合計6通りの読み取られ方が考えられます。この6通りの読み取られ方を読み枠と言います。あるいはリーディングフレームとも言います。6個の読み枠のうち、タンパク質をコードを指定している読み枠は1個だけです。
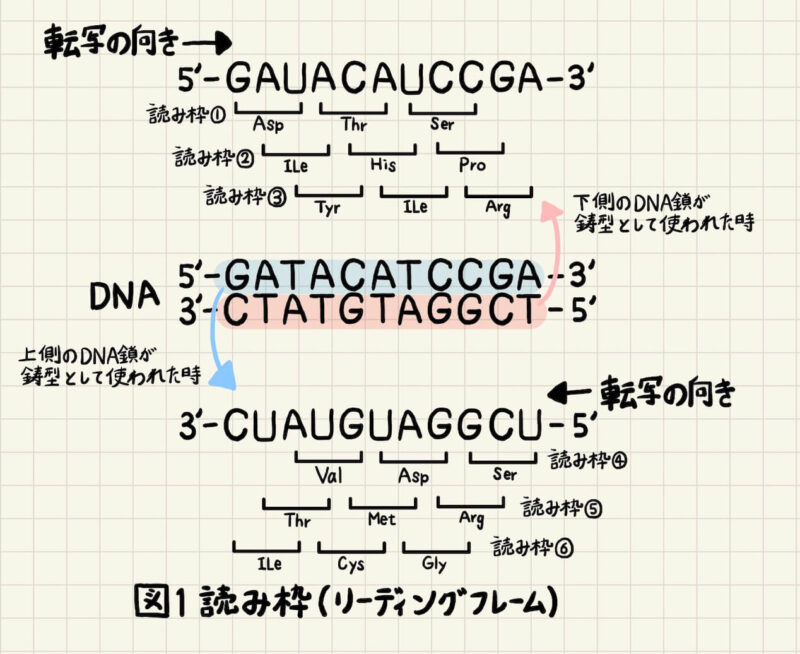
ゲノムを調べてみると、終始コドンがなく、アミノ酸を指定するコドンが長く続いている読み枠を見つけることができます。このようなDNA領域をオープンリーディングフレーム(open reading frame; ORF)と言います。ORFはタンパク質をコードしている可能性が高く、ゲノムから遺伝子を推定するのに役立ちます。

読み枠が1塩基ずれるだけで、全く異なるタンパク質ができてしまいます。
医学研究において、生物や培養細胞中の遺伝子の読み枠を意図的にずらし、特定の遺伝子の機能を完全に除去する手法があります。このように遺伝子の機能を完全に無くすことをノックアウト(Knockout; KO)と言いますが、遺伝子のKOによりその遺伝子の機能を調べることができます。
ORFとよく似た意味の用語で、CDS(Coding Sequence)があります。研究の現場ではCDSもよく出てくるのでORFとの違いを説明しておきます。CDSは実際にタンパク質に翻訳されるDNA領域またはRNA領域のことを言い、イントロンが含まれておらずエキソンのみで構成されています。また、CDSは開始コドンで始まり、終始コドンで終わります。一方で、ORFも開始コドンで始まり、終始コドンで終わりますが、イントロンも含むDNA領域を指しています。
tRNAがアミノ酸を運ぶ
タンパク質の合成は、表1に示したように、それぞれのコドンに適切なアミノ酸が運ばれる必要があります。mRNAのコドンから適切なアミノ酸が選ばれる仕組みについて見ていきましょう。
アミノアシルtRNA
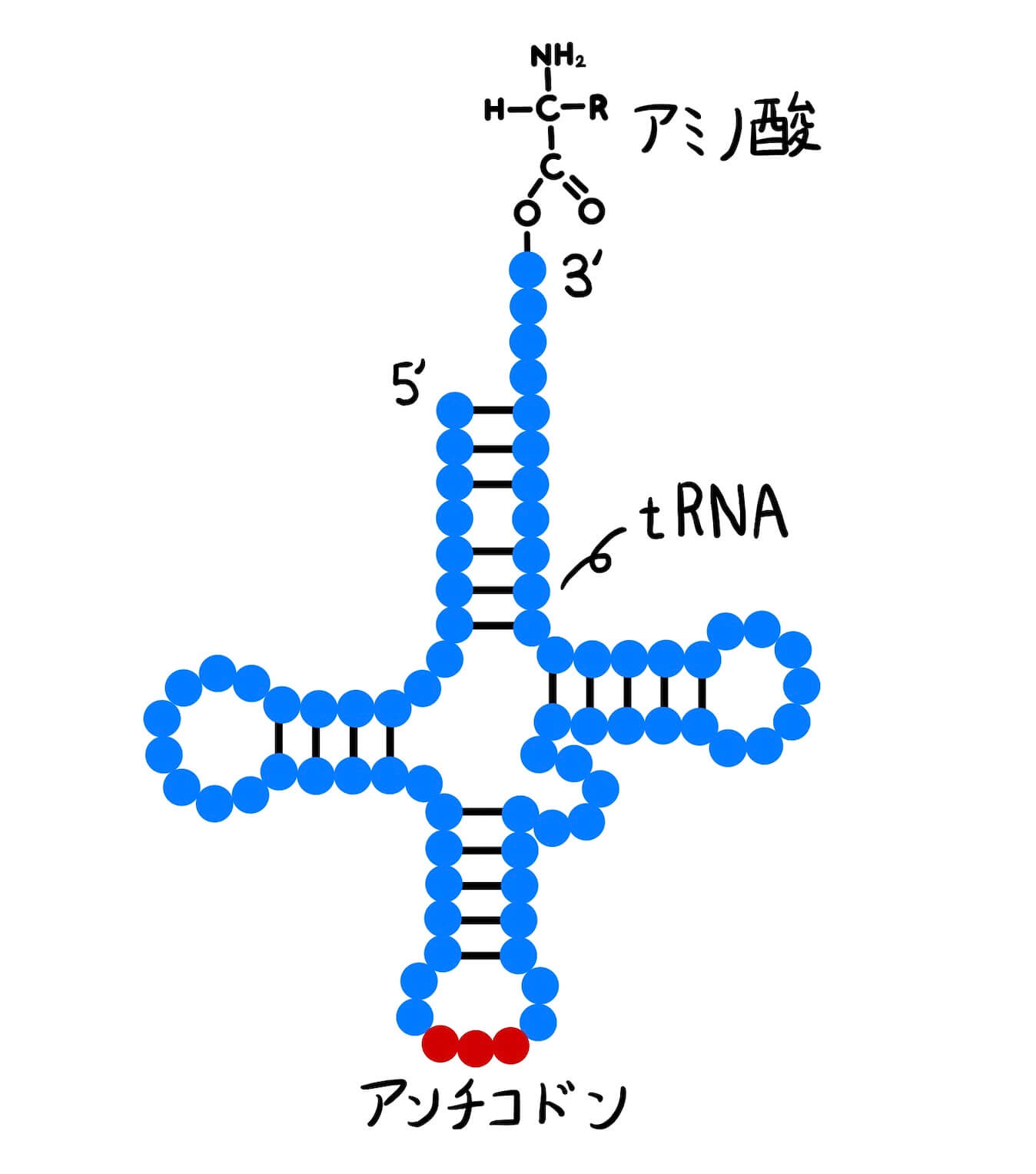
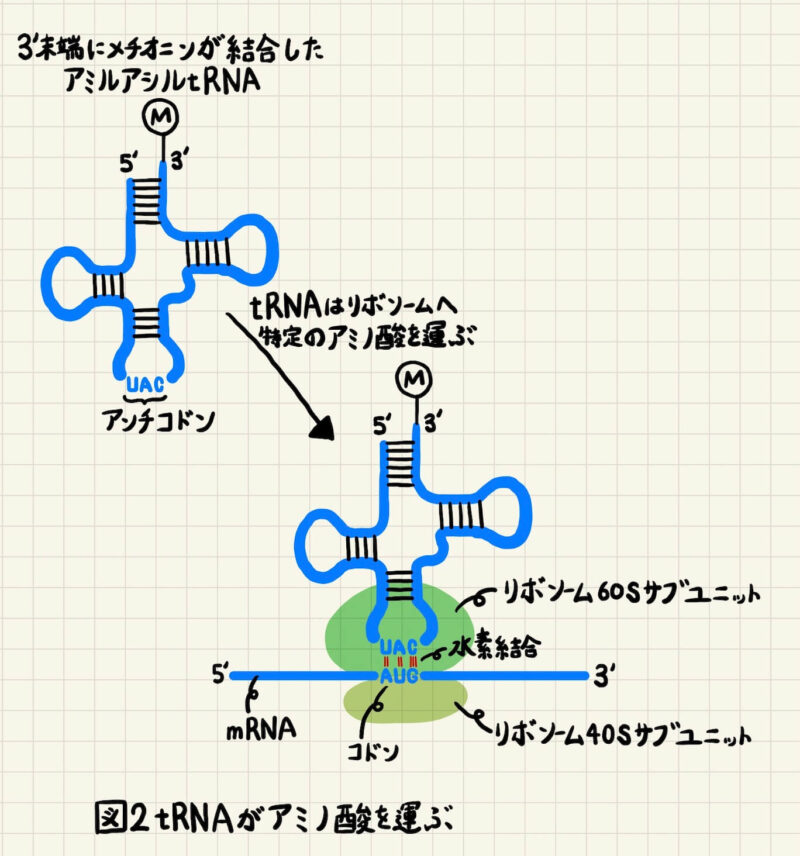
適切なコドンへの適切なアミノ酸の運搬は、transfer RNA(tRNA)という非翻訳RNAが担っています。 tRNAは三つ葉のクローバーのような構造をしており、3’末端にアミノ酸を結合してコドンまで運びます(図2)。 図2のようにアミノ酸と結合したtRNAをアミノアシルtRNAと言います。
また、tRNA中にあるコドンを認識する連続した3塩基をアンチコドンと言います。 アンチコドンは適切なコドンと相補的な塩基対を形成するので、tRNAはアミノ酸を適切なコドンまで運ぶことができます。 図2では、例としてメチオニン(Met, M)と結合したtRNAを描いています。アンチコドンはUACで、メチオニンを指定するコドンのAUGと結合できるので、正しくメチオニンを運搬していることが分かります。例としてメチオニンを運搬するtRNAを描きましたが、アミノ酸は20種類あるので、tRNAもそれぞれのアミノ酸に対応したものが存在しています。
tRNAのアミノアシル化
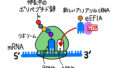
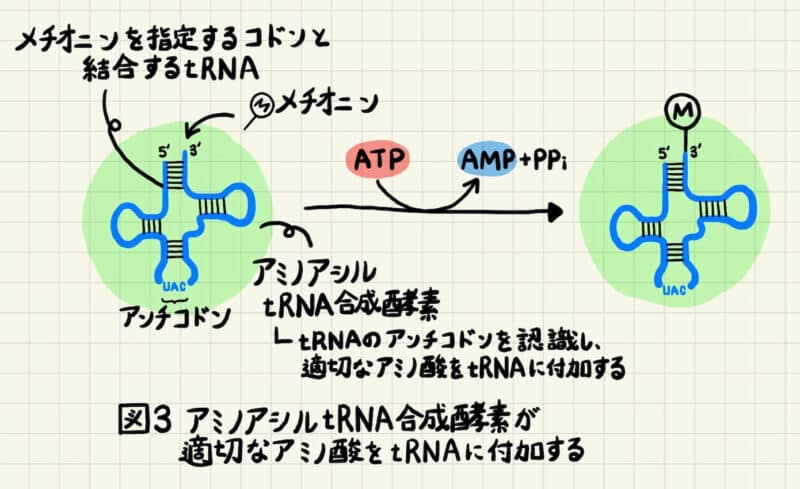
前述した通り、tRNAは20種類のアミノ酸を適切なコドンまで運搬する役割を持っており、20種類のアミノ酸それぞれに対応する特異的なtRNAが存在しています。tRNAのアンチコドンがどのアミノ酸を指定しているかを認識し、tRNAに適切なアミノ酸を付加する酵素をアミノアシルtRNA合成酵素と言います(図3)。アミノアシルtRNA合成酵素も20種類のアミノ酸それぞれに対応した特異的な酵素が存在しています。 図3の例は、メチオニン専用のアミノアシルtRNA合成酵素がメチオニン専用のtRNAと出会い、そのアンチコドンが本当にメチオニンを指定しているのかを確認してから適切なアミノ酸(この場合はメチオニン)を付加している場面を描いています。
まとめると、あるコドンと結合する特定のtRNAは、特定のアミノアシルtRNA合成酵素によって正しいアミノ酸が付加され、mRNAの遺伝情報はアミノ酸へと移っていきます。
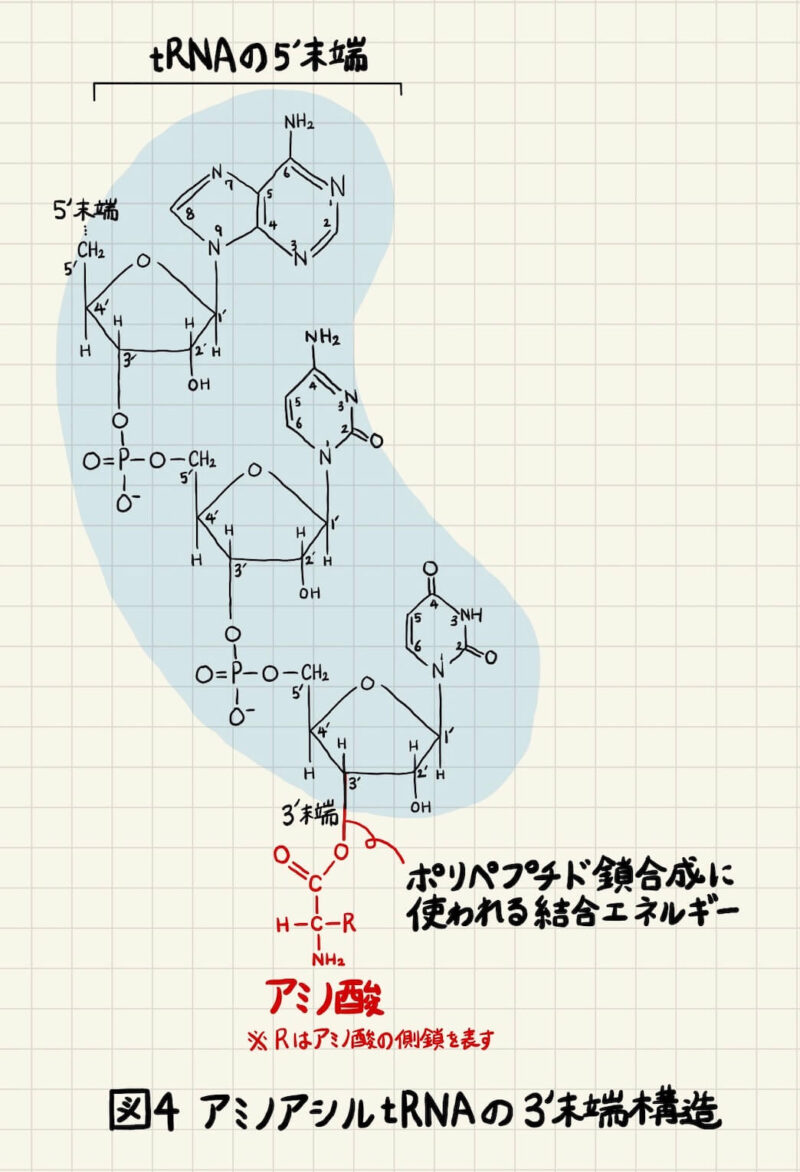
続いてtRNAのアミノアシル化の分子機構をもう少し詳しく見ていきます。図4のようにアミノ酸はtRNAの3’末端に付加されます。図3中に描いたのですが、アミノアシルtRNA合成酵素によるtRNAのアミノアシル化にはATPエネルギーが必要です。ATPはこの反応でAMPとピロリン酸(PPi)となり、ATPに蓄えられていたエネルギーはtRNAとアミノ酸の結合エネルギーに変えられます(図4)。この結合エネルギーはポリペプチド鎖が伸長される時(=タンパク質の合成)に使われます。
これまで学んできたように、アミノアシルtRNA合成酵素はtRNAとアミノ酸の正しい組み合わせの時にのみ、tRNAのアミノアシル化を触媒する機能を持ちますが、そのほかに校正機能も備えています。すなわち、アミノアシルtRNA合成酵素が誤ったアミノ酸を付加してしまった場合、自身が持つ加水分解作用により、誤ったアミノ酸をtRNAから取り除きます。
まとめ
- アミノ酸を指定するmRNAの連続した3塩基をコドンという。
- 開始コドンは「AUG」、終始コドンは「UAA」、「UAG」、「UGA」の3種類。
- 6個の読み枠のうち、タンパク質をコードを指定している読み枠は1個だけ。
- 終始コドンがなく、アミノ酸を指定するコドンが続く読み枠をオープンリーディングフレーム(open reading frame; ORF)という。
- コドンを認識するtRNA中の連続した3塩基をアンチコドンという。
- 特異的なアミノアシルtRNA合成酵素が、tRNAに適切なアミノ酸を付加する。
- tRNAの3’末端にアミノ酸が結合したものをアミノアシルtRNAといい、アミノ酸を運搬する。