はじめに
あるコドンと結合する特定のtRNAは、特定のアミノアシルtRNA合成酵素によって正しいアミノ酸が付加され、mRNAの遺伝情報はアミノ酸配列へと変換されます。このtRNAとアミノ酸の特異的な結合の仕組みについては、以下の記事で詳しく解説しました。今回は、アミノ酸と結合したtRNA(=アミノアシルtRNA)がリボソームに運搬され、アミノ酸が重合されていく段階について詳しく解説していきます!今回の記事は、以下のtRNAとアミノ酸の記事を理解していれば、比較的簡単であると思います。
みんなで学んでいきましょう!
※tRNAとアミノ酸についての解説は以下の記事をお読みください。
※翻訳の開始機構については以下の記事で詳しく解説しています。
【難易度】★☆☆☆☆
【重要度】★★★★★
リボソームの構造
タンパク質はアミノ酸の重合体であり、タンパク質の合成とはアミノ酸とアミノ酸をつなげる反応のことをいいます。このアミノ酸をつなげてタンパク合成を行う酵素がリボソームです。リボソームはrRNAとタンパク質の複合体で、リボソームの約2/3がrRNA、約1/3がタンパク質でできています。
リボソームは大サブユニットと小サブユニットで構成されています(図1)。大サブユニットと小サブユニットは核内で組み立てられた後、それぞれ細胞質に移動し、細胞質で合体してタンパク合成を行うことのできる機能的なリボソームになります。 真核生物のリボソームにはtRNAとの結合部位が2つ(A部位、P部位)と、mRNAとの結合部位が1つあります。ただし、大腸菌の場合はtRNAとの結合部位としてE部位もあります。
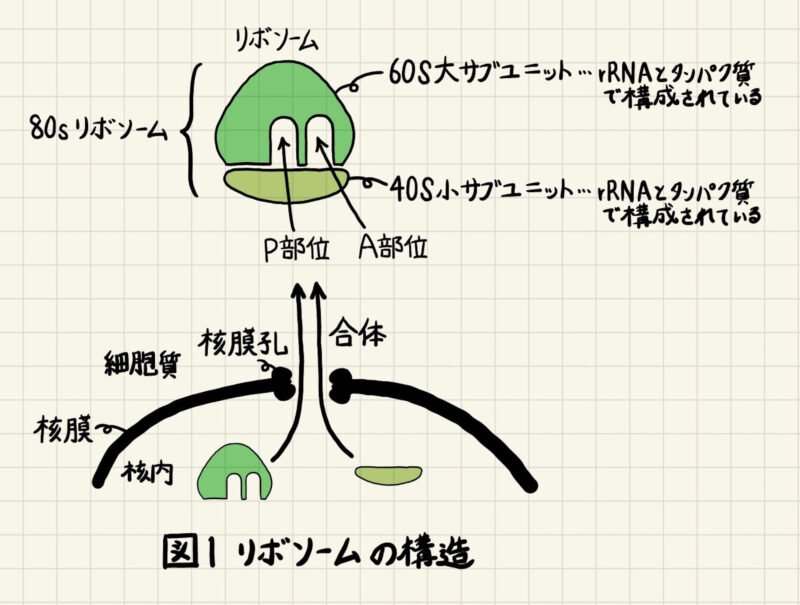
サブユニットの名前は、超遠心をかけたときの沈降速度(Svedberg; S)を用いて表されます。 沈降速度Sは一般的にリボソームを表す時にのみ使われます。 Sの前につく数字が大きいほど沈降速度が速く、rRNAも大きくなります。真核生物のリボソームは60S大サブユニットと40S小サブユニットで構成されていますが、原核生物のリボソームは50S大サブユニットと30S小サブユニットで構成されています。図1中の60S大サブユニットと40S小サブユニットの沈降係数の合計が100Sとならず80Sとなっているのは、沈降係数はサイズだけでなく形状にも依存するからです。

ほとんど全ての細胞で一定量の発現があり、細胞の維持や増殖に欠かせない遺伝子のことをハウスキーピング遺伝子と言います。rRNA、GAPDH、β-Actinなどがハウスキーピング遺伝子として古くから有名です。
翻訳の概要
タンパク質の合成は、翻訳開始を意味する開始コドン「AUG」から始まります。この開始コドンはmRNAの5’末端にあるのではなく、端から数十塩基離れた位置に存在しています(図2)。終始コドンも同様で、mRNAの末端にあるのではなく、3’末端からは少し離れた位置に存在しています。すなわち、タンパク質に翻訳されるのはmRNAの中央部分だけであり、両端は翻訳されません。この非翻訳領域をUTR(untranslated region)といい、図2のようにmRNAの5’側のUTRを5’UTR、3’側のUTRを3’UTRと呼びます。このUTRは生物学的にとても重要で、5’UTRはmRNAの転写量調節機能があり、3’UTRはmRNAの細胞質での局在(=位置)を決めます。
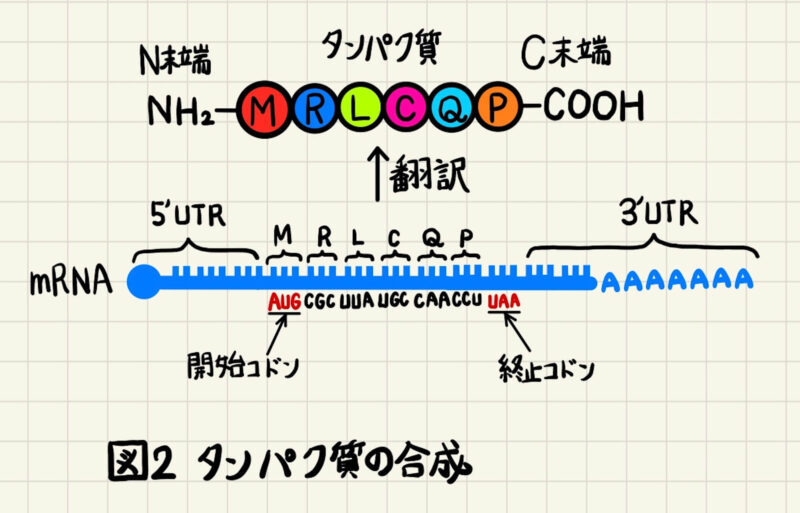

図1は紙面の都合上、mRNAやタンパク質を短く書いていますが、実際はもっとサイズが大きい点に注意してください!
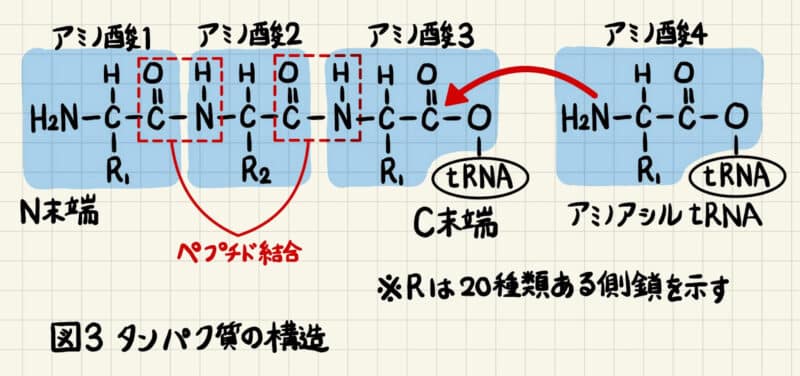
また、図2に図示したようにタンパク質の末端はN末端とC末端と呼ばれています。図3に翻訳中のタンパク質の分子構造を描きました。タンパク質はアミノ酸同士がペプチド結合という結合でつながっています。ペプチド鎖のカルボキシ基(-COOH)に新しいアミノ酸のアミノ基(-NH2)が付加されることでタンパク質は伸長していきます。つまりタンパク質の端はアミノ基とカルボキシ基であり、それぞれN末端、C末端と言います。DNA複製や転写の合成方向が5’→3’と決まっていたように、タンパク質合成もN末端→C末端と合成の方向が決まっています。通常N末端を左側、C末端を右側にして書きます。
ポリペプチド鎖の伸長
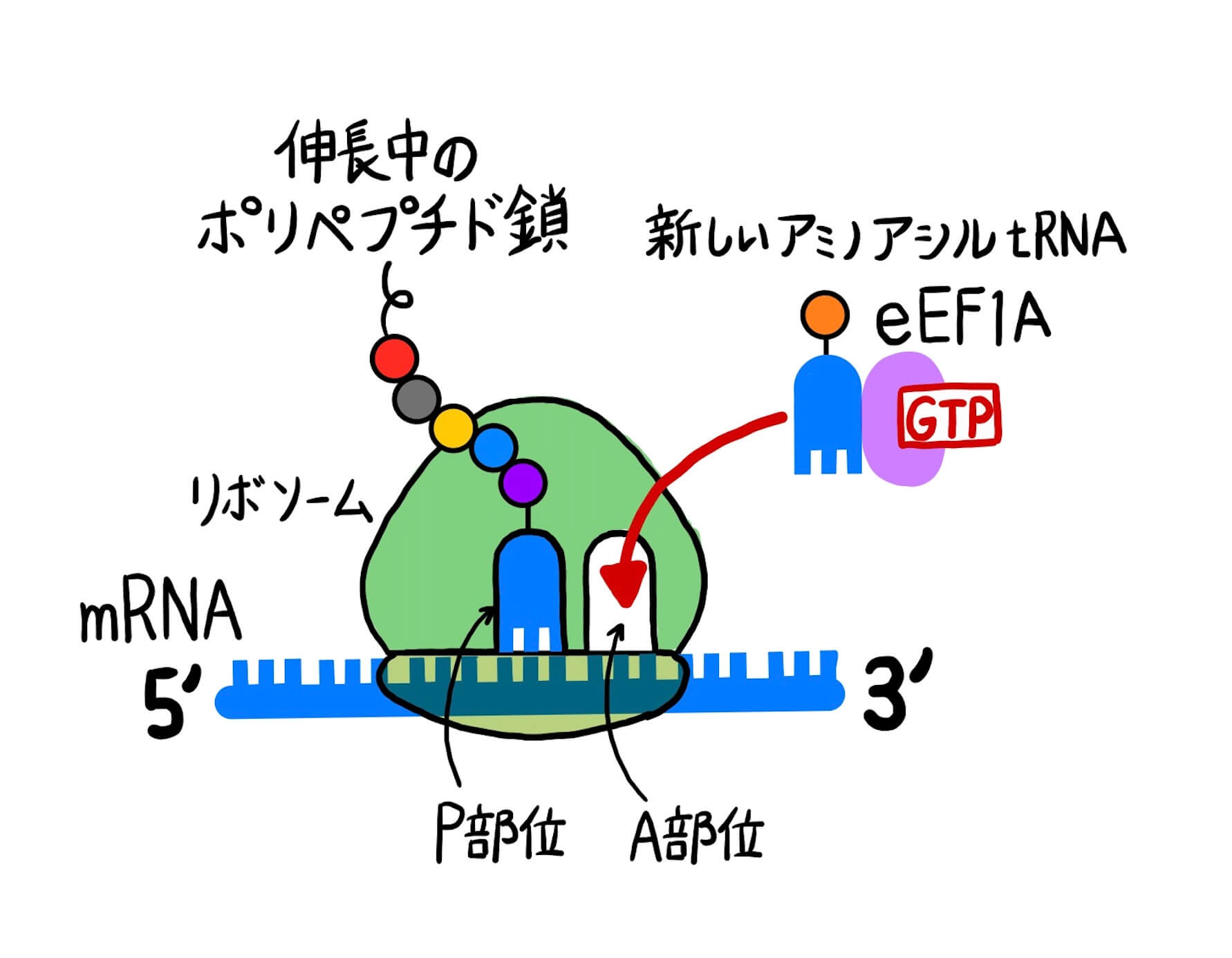
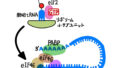
ポリペプチド鎖の伸長はまず、伸長因子eET1A(eukaryotic translation elongation factor 1 alpha 1)と結合したアミノアシルtRNAがリボソームのA部位に運ばれます(図4①)。伸長因子eET1AはアミノアシルtRNAのリボソームへの結合を触媒しています。アミノアシルtRNAのアンチコドンとmRNAのコドンが適合している場合は、伸長因子eET1Aが結合しているGTPがGDPに加水分解され、アミノアシルtRNAから解離します(図4②)。細菌ではこの伸長因子はEF-Tuと呼ばれます。
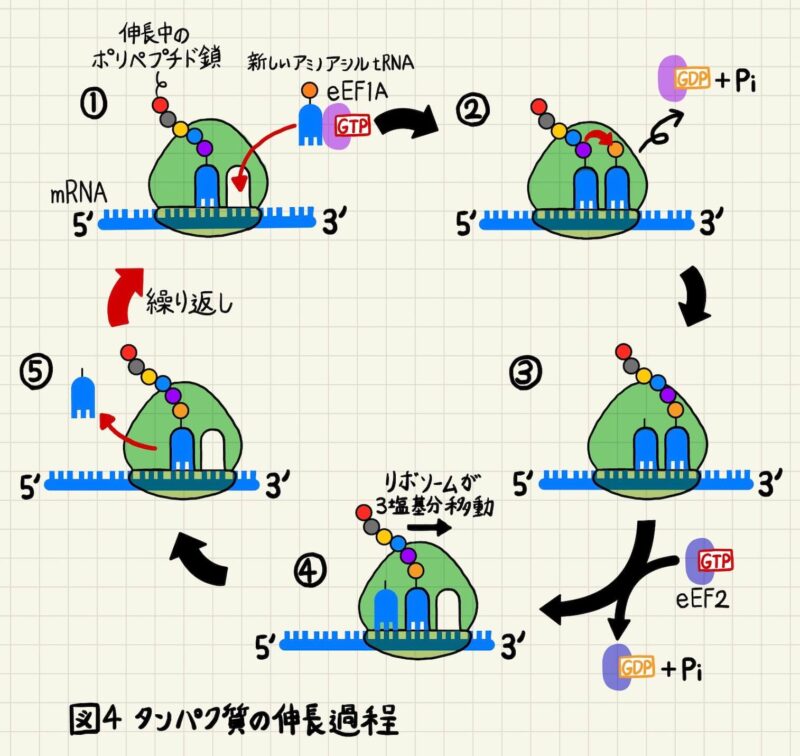
次に、60S大サブユニットのペプチジルトランスフェラーゼ活性(ペプチジル基転移活性)により、P部位にあるポリペプチド鎖がA部位にあるアミノ酸のアミノ基に転移されます(図4③)。その後、リボソームがmRNAの3’末端方向に3塩基分(=1コドン分)移動します(図4④)。このリボソームの移動にはGTPの結合したeEF2の加水分解が必要です。細菌ではこの伸長酵素はEF-Gと呼ばれます。
リボソームの移動により、ポリペプチド鎖とつながったtRNAはA部位からP部位へ移動したことになり、A部位は空の状態になります。また、このリボソームの移動により、P部位のtRNAはリボソームから解離します(図4⑤)。すると、新たに空いたA部位に新しいアミノアシルtRNAの受け入れが可能になり、ペプチド鎖の伸長サイクルが図4の①に戻ります。これを繰り返してタンパク質の合成は行われます。
また、DNAポリメラーゼやアミノアシルtRNA合成酵素などと同様に、リボソームも校正機能を有しており、高い正確性で翻訳を行うことが可能です。
抗生物質
細菌などの微生物の増殖を阻害する物質を抗生物質と言います。抗生物質の多くは、細菌とヒトのリボソームの構造の違いを利用して、細菌のリボソームのみを阻害します。 タンパク質は全ての生物の主要な構成要素であり、タンパク質の合成を行うリボソームは生命維持に必須の酵素です。そのため抗生物質によりタンパク合成を阻害された細菌は生きていくことができません。
表1に有名な抗生物質を示しました。真核生物のリボソームに効く抗生物質は薬には使えませんが、タンパク質合成阻害試薬に頻用されます。

抗生物質に耐性を持つ薬剤耐性菌
最後に、薬剤耐性菌問題について取り上げます。これは人類全員にとって非常に重要な問題であるので、ここで正しい知識を学んでいきましょう!
世界的に増加する薬剤耐性菌
近年、抗生物質の効かない薬剤耐性菌(Antimicrobial Resistance; AMR)が世界中で増えてきています。薬剤耐性菌が増加すると、抗生物質が効かなくなり、細菌感染症に対してなすすべがなくなってしまいます。実際に、薬剤耐性菌が原因で亡くなった人の数は2013年で70万人を超え、このまま何も対策を取らない場合、その死者数は2050年に1,000万人を上回ると予想されています。したがって薬剤耐性菌の拡大防止は、人類が今後も地球上で反映していくためにとても重要なことです。
薬剤耐性菌を拡大させないために
細菌が抗生物質に出会った時、薬剤に感受性の高い細菌から順番に死んでいきます。したがって、 自己判断で抗生物質の服用回数を減らしたり、途中で服用を中断してしまったりすると、細菌を全滅させることができず、耐性菌出現の可能性を高めてしまいます。病院で抗生物質を処方された場合は、医師や薬剤師の指示を守って適切に使用し、飲み切ることが重要です。
また、抗生物質をむやみに使用しないということも大切です。通常の風邪や、インフルエンザ、新型コロナウイルスはウイルス感染であり、抗生物質は効きません。したがって、医師に自分の症状をできるだけ正確に説明し、抗生物質が必要でない場合は、使用しないという選択をしましょう。
まとめ
- リボソームは大サブユニットと小サブユニットで構成される。
- 真核生物のリボソームにはtRNAとの結合部位が2つ(A部位、P部位)と、mRNAとの結合部位が1つある。
- リボソームはP部位のペプチド鎖とA部位のアミノ酸の結合を触媒し、タンパク質を合成していく。
- タンパク質に翻訳されないmRNAの末端領域をUTRをいう。
- 抗生物質の多くは細菌のリボソームのみを阻害して、細菌の増殖を抑制する。